热带海洋学报 ›› 2024, Vol. 43 ›› Issue (6): 145-159.doi: 10.11978/2023196CSTR: 32234.14.2023196
食蟹豆齿鳗线粒体基因组结构特征及系统进化分析
- 浙江海洋大学水产学院, 浙江 舟山 316022
-
收稿日期:2023-12-15修回日期:2024-02-11出版日期:2024-11-10发布日期:2024-12-05 -
通讯作者:杨天燕 -
作者简介:杨澜(2001—), 女, 浙江省衢州市人, 研究方向为海洋生物资源与环境。email: 3559892237@qq.com
-
基金资助:浙江省重点研发计划项目(2021C2047); 舟山市科技计划项目(2022C41022); 国家级大学生创新创业训练计划项目(202310340056); 浙江省大学生科技创新活动计划暨新苗人才计划项目(2023R411005); 浙江海洋大学校级大学生科研创新计划项目(2023-A-025)
The mitochondrial genomic characteristics and phylogenetic relationship analysis of Pisodonophis cancrivorus (Anguilliformes, Ophichthidae)
YANG Lan( ), ZHAO Yao, LIU Yuping, YANG Tianyan(
), ZHAO Yao, LIU Yuping, YANG Tianyan( )
)
- School of Fisheries, Zhejiang Ocean University, Zhoushan 316022, China
-
Received:2023-12-15Revised:2024-02-11Online:2024-11-10Published:2024-12-05 -
Contact:YANG Tianyan -
Supported by:Province Key Research and Development Program of Zhejiang(2021C2047); Technology Planning Project of Zhoushan(2022C41022); National Innovation and Entrepreneurship Training Program for College Students(202310340056); Science and Technology Innovation Project of College Students in Zhejiang Province(2023R411005); Innovation Project of College Students in Zhejiang Ocean University(2023-A-025)
摘要:
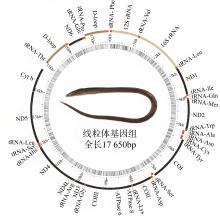
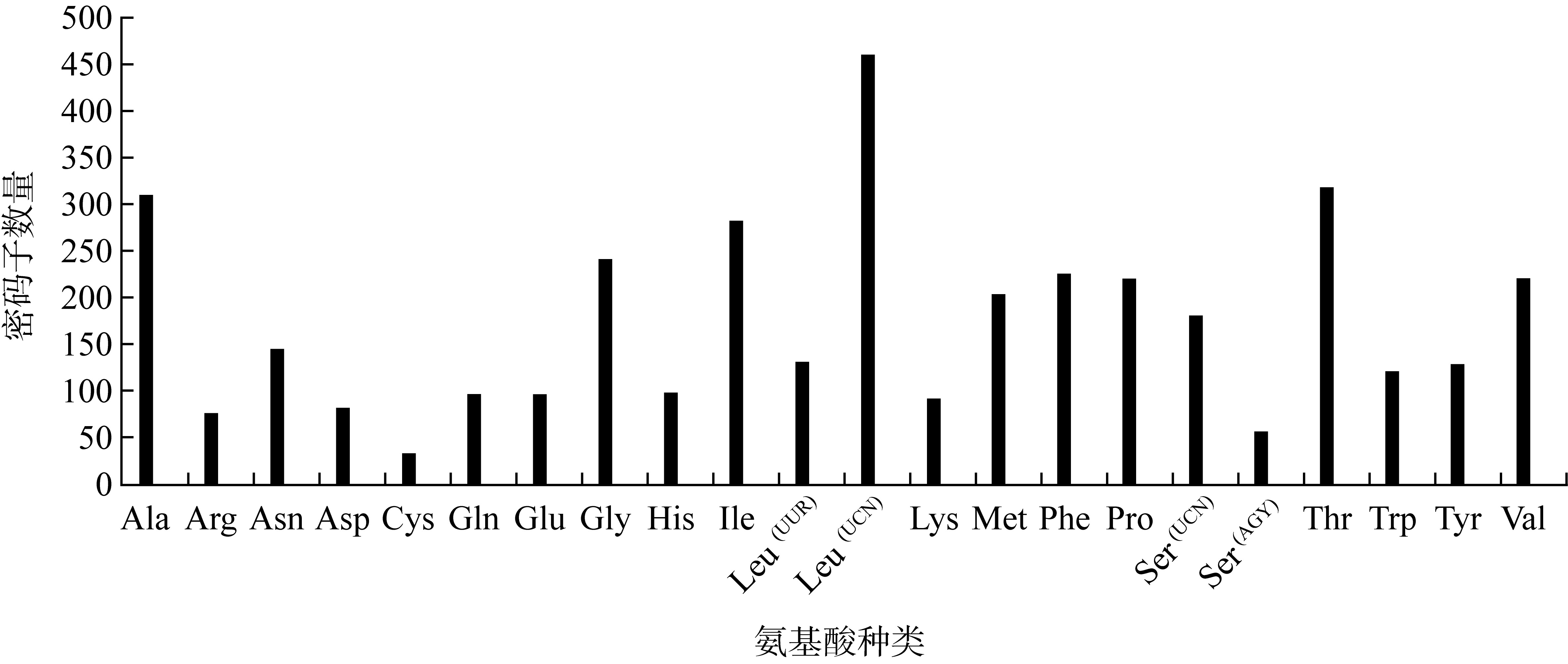
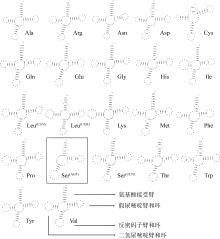
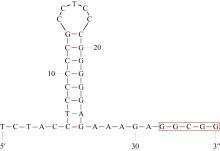
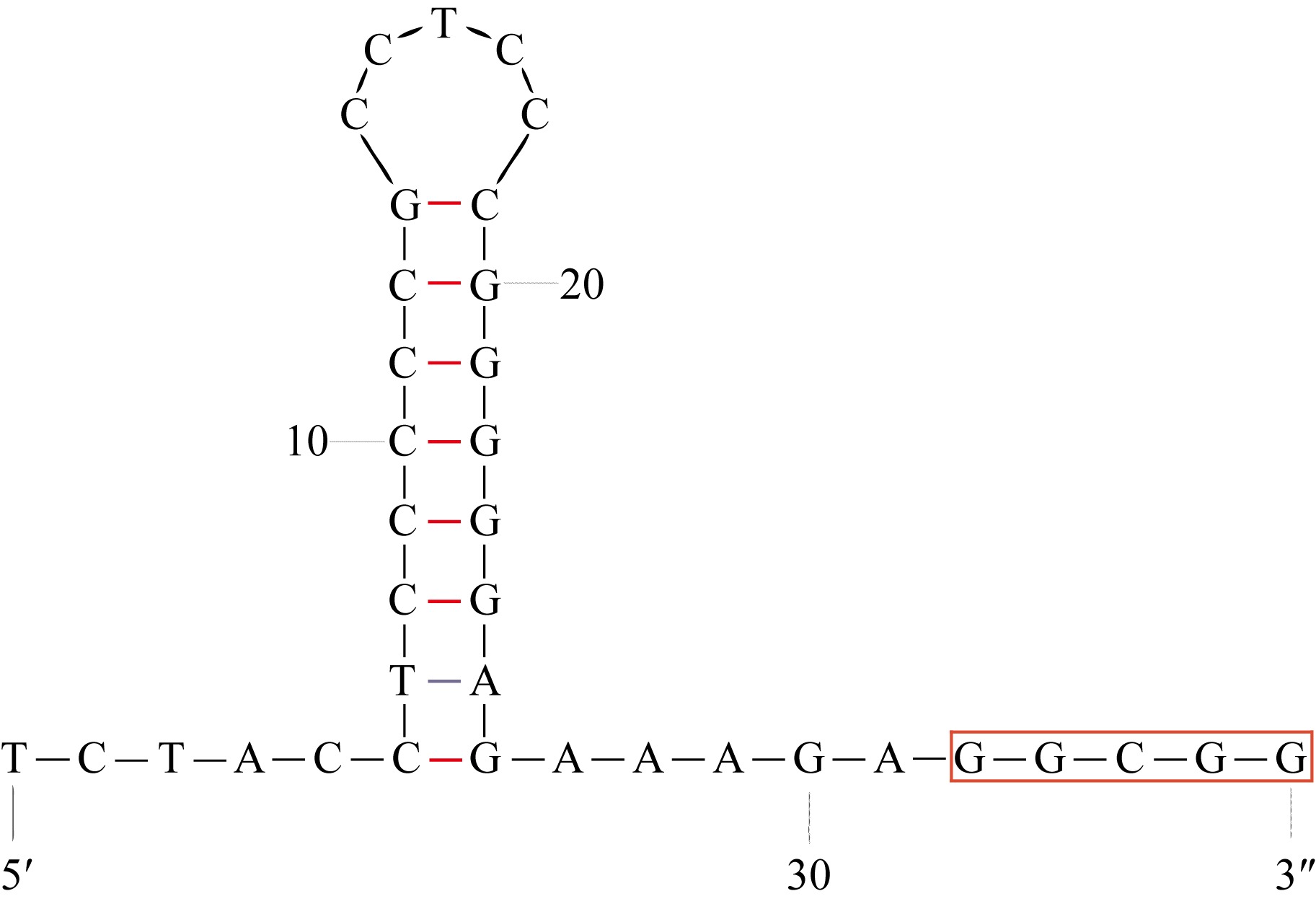
食蟹豆齿鳗(Pisodonophis cancrivorus)是我国东南沿海重要的渔获种类之一。本研究采用高通量测序技术对采自于舟山近海的食蟹豆齿鳗线粒体基因组进行了测定, 并对其结构特征及系统发育关系进行了分析。食蟹豆齿鳗线粒体DNA序列全长为17650bp, 4种碱基含量分别为A(31.72%)、G(15.58%)、T(26.26%)和C(26.44%), 表现出明显的反G偏倚和A+T富集现象。与已发表的蛇鳗科鱼类相似, 其线粒体部分基因由于自然选择的作用, 发生了串联复制-随机丢失(tandem duplication-random loss, TDRL)事件, 导致ND6基因和tRNA-Glu(E)转移到tRNA-Thr(T)和tRNA-Pro(P)之间, 并且ND6基因上游出现了另一个长度为966bp的控制区, 两个控制区呈现独立演化的特征。13个蛋白质编码基因中分别存在2种启动子(ATG、GTG)和4种终止子(TAG、TAA、TA-、T--), 且仅COI基因以GTG作为起始密码子。编码的3 824个氨基酸中, 亮氨酸(Leu)含量最高而半胱氨酸(Cys)含量最低。22种tRNA的二级结构中存在17处碱基错配, 且仅tRNA-SerAGY(S)由于缺少二氢尿嘧啶臂(DHU臂)无法折叠形成典型的三叶草结构。长度为21bp的轻链复制起始区(origin of L-strand replication region, OL)位于“WANCY”基因簇内, 其发夹型二级结构(hairpin secondary structure)的3'端具有与爬行动物类似的保守功能基序3'-GGCGG-5'。将17种蛇鳗科鱼类的线粒体12个蛋白质编码基因序列去除终止密码子之后进行串联, 基于Kimura双参数模型计算种间遗传距离为0.128~0.297。使用最大似然法(maximum likelihood, ML)和贝叶斯法(Bayesian inference, BI)构建系统发育树, 显示了蛇鳗科鱼类复杂的演化关系, 食蟹豆齿鳗位于进化树的基部, 与短尾蛇鳗(Ophichthus brevicaudatus)和艾氏蛇鳗(O.evermanni)亲缘关系最近, 是该类群中较晚发生遗传分化的种类。
中图分类号:
- S917.4
引用本文
杨澜, 赵耀, 刘玉萍, 杨天燕. 食蟹豆齿鳗线粒体基因组结构特征及系统进化分析[J]. 热带海洋学报, 2024, 43(6): 145-159.
YANG Lan, ZHAO Yao, LIU Yuping, YANG Tianyan. The mitochondrial genomic characteristics and phylogenetic relationship analysis of Pisodonophis cancrivorus (Anguilliformes, Ophichthidae)[J]. Journal of Tropical Oceanography, 2024, 43(6): 145-159.
表1
食蟹豆齿鳗线粒体基因组注释"
| 基因 | 编码链 | 起始位点 | 终止位点 | 长度/bp | 基因间隔/bp | 编码氨基酸数量 | 起始密码子 | 终止密码子 | 反密码子 |
|---|---|---|---|---|---|---|---|---|---|
| tRNA-Phe(F) | H | 1 | 68 | 68 | 0 | GAA | |||
| 12S rRNA | H | 69 | 1 027 | 959 | 0 | ||||
| tRNA-Val(V) | H | 1 028 | 1 098 | 71 | 0 | TAC | |||
| 16S rRNA | H | 1 099 | 2 799 | 1 701 | 0 | ||||
| tRNA-LeuUUR(L) | H | 2 800 | 2 875 | 76 | 0 | TAA | |||
| ND1 | H | 2 876 | 3 844 | 969 | 0 | 322 | ATG | TAA | |
| tRNA-Ile(I) | H | 3 847 | 3 919 | 73 | 2 | GAT | |||
| tRNA-Gln(Q) | L | 3 919 | 3 989 | 71 | -1 | TTG | |||
| tRNA-Met(M) | H | 3 989 | 4 057 | 69 | -1 | CAT | |||
| ND2 | H | 4 058 | 5 114 | 1 057 | 0 | 352 | ATG | T-- | |
| tRNA-Trp(W) | H | 5 115 | 5 183 | 69 | 0 | TCA | |||
| tRNA-Ala(A) | L | 5 185 | 5 253 | 69 | 1 | TGC | |||
| tRNA-Asn(N) | L | 5 255 | 5 327 | 73 | 1 | GTT | |||
| OL | H | 5 329 | 5 349 | 21 | 1 | ||||
| tRNA-Cys(C) | L | 5 371 | 5 437 | 67 | 21 | GCA | |||
| tRNA-Tyr(Y) | L | 5 438 | 5 509 | 72 | 0 | GTA | |||
| COI | H | 5 511 | 7 151 | 1 641 | 1 | 546 | GTG | TAA | |
| tRNA-SerUCN(S) | L | 7 169 | 7 239 | 71 | 17 | TGA | |||
| tRNA-Asp(D) | H | 7 245 | 7 312 | 68 | 5 | GTC | |||
| COII | H | 7 319 | 8 009 | 691 | 6 | 230 | ATG | T-- | |
| tRNA-Lys(K) | H | 8 010 | 8 084 | 75 | 0 | TTT | |||
| ATP8 | H | 8 086 | 8 253 | 168 | 1 | 55 | ATG | TAA | |
| ATP6 | H | 8 244 | 8 923 | 680 | -10 | 226 | ATG | TA- | |
| COIII | H | 8 924 | 9 708 | 785 | 0 | 261 | ATG | TA- | |
| tRNA-Gly(G) | H | 9 709 | 9 780 | 72 | 0 | TCC | |||
| ND3 | H | 9 781 | 10 129 | 349 | 0 | 116 | ATG | T-- | |
| tRNA-Arg(R) | H | 10 130 | 10 199 | 70 | 0 | TCG | |||
| ND4L | H | 10 200 | 10 496 | 297 | 0 | 98 | ATG | TAA | |
| ND4 | H | 10 490 | 11 870 | 1 381 | -7 | 610 | ATG | T-- | |
| tRNA-His(H) | H | 11 871 | 11 939 | 69 | 0 | GTG | |||
| tRNA-SerAGY(S) | H | 11 940 | 12 009 | 70 | 0 | GCT | |||
| tRNA-LeuCUN(L) | H | 12 010 | 12 082 | 73 | 0 | TAG | |||
| ND5 | H | 12 083 | 13 918 | 1 836 | 0 | 611 | ATG | TAG | |
| Cyt b | H | 13 933 | 15 073 | 1 141 | 14 | 380 | ATG | T-- | |
| tRNA-Thr(T) | H | 15 074 | 15 145 | 72 | 0 | TGT | |||
| D-loop1 | H | 15 146 | 16 111 | 966 | 0 | ||||
| ND6 | L | 16 112 | 16 630 | 519 | 0 | 172 | ATG | TAG | |
| tRNA-Glu(E) | L | 16 631 | 16 699 | 69 | 0 | TTC | |||
| tRNA-Pro(P) | L | 16 703 | 16 775 | 73 | 3 | TGG | |||
| D-loop2 | H | 16 776 | 17 650 | 875 | 0 |
表2
食蟹豆齿鳗线粒体碱基组成与偏倚"
| 基因/区域 | 长度/bp | A/% | T/% | G/% | C/% | A+T/% | AT偏倚 | GC偏倚 |
|---|---|---|---|---|---|---|---|---|
| 线粒体全序列 | 17 650 | 31.72 | 26.26 | 15.58 | 26.44 | 57.98 | 0.094 | -0.259 |
| 13个蛋白质编码基因 | 11 514 | 29.33 | 28.23 | 15.46 | 26.98 | 57.56 | 0.019 | -0.271 |
| 密码子第1位点 | 3 849 | 28.55 | 23.33 | 22.79 | 25.33 | 51.88 | 0.101 | -0.053 |
| 密码子第2位点 | 3 831 | 23.52 | 35.47 | 13.94 | 27.07 | 58.99 | -0.203 | -0.320 |
| 密码子第3位点 | 3 834 | 35.92 | 25.93 | 9.62 | 28.53 | 61.85 | 0.162 | -0.496 |
| ND1 | 969 | 27.86 | 27.86 | 15.38 | 28.90 | 55.72 | 0.000 | -0.305 |
| ND2 | 1 057 | 33.59 | 24.50 | 13.62 | 28.29 | 58.09 | 0.156 | -0.350 |
| COI | 1 641 | 28.28 | 29.25 | 17.55 | 24.92 | 57.53 | -0.017 | -0.174 |
| COII | 691 | 30.82 | 26.19 | 15.63 | 27.36 | 57.01 | 0.081 | -0.273 |
| ATP8 | 168 | 33.33 | 25.00 | 10.12 | 31.55 | 58.33 | 0.143 | -0.514 |
| ATP6 | 680 | 29.12 | 28.97 | 12.35 | 29.56 | 58.09 | 0.003 | -0.411 |
| COIII | 785 | 28.15 | 27.90 | 17.20 | 26.75 | 56.05 | 0.004 | -0.217 |
| ND3 | 349 | 28.37 | 31.52 | 14.04 | 26.07 | 59.89 | -0.053 | -0.300 |
| ND4L | 297 | 28.28 | 26.60 | 13.47 | 31.65 | 54.88 | 0.031 | -0.403 |
| ND4 | 1 381 | 29.98 | 27.59 | 13.76 | 28.67 | 57.57 | 0.042 | -0.351 |
| ND5 | 1 836 | 32.57 | 26.85 | 12.91 | 27.67 | 59.42 | 0.096 | -0.364 |
| Cyt b | 1 141 | 28.66 | 29.71 | 15.07 | 26.56 | 58.37 | -0.018 | -0.276 |
| ND6 | 519 | 38.73 | 15.03 | 14.06 | 32.18 | 53.76 | 0.441 | -0.392 |
| 12S rRNA | 959 | 33.58 | 21.38 | 20.02 | 25.02 | 54.96 | 0.222 | -0.111 |
| 16S rRNA | 1 701 | 37.92 | 19.05 | 18.99 | 24.04 | 56.97 | 0.331 | -0.117 |
| D-loop1 | 966 | 32.19 | 32.51 | 14.29 | 21.01 | 64.70 | 0.441 | -0.392 |
| D-loop2 | 875 | 36.23 | 29.60 | 10.74 | 23.43 | 65.83 | 0.441 | -0.392 |
表3
13个蛋白质编码基因密码子使用频率"
| 密码子 | 计数 | RSCU | 密码子 | 计数 | RSCU | 密码子 | 计数 | RSCU | 密码子 | 计数 | RSCU |
|---|---|---|---|---|---|---|---|---|---|---|---|
| UUU(F) | 121 | 1.07 | UCU(S) | 30 | 0.76 | UAU(Y) | 62 | 0.96 | UGU(C) | 10 | 0.61 |
| UUC(F) | 105 | 0.93 | UCC(S) | 63 | 1.59 | UAC(Y) | 67 | 1.04 | UGC(C) | 23 | 1.39 |
| UUA(L) | 101 | 1.02 | UCA(S) | 84 | 2.12 | UAA(*) | 0 | 0.00 | UGA(W) | 108 | 1.79 |
| UUG(L) | 31 | 0.31 | UCG(S) | 4 | 0.10 | UAG(*) | 1 | 0.80 | UGG(W) | 13 | 0.21 |
| CUU(L) | 108 | 1.09 | CCU(P) | 32 | 0.58 | CAU(H) | 33 | 0.67 | CGU(R) | 11 | 0.58 |
| CUC(L) | 108 | 1.09 | CCC(P) | 67 | 1.22 | CAC(H) | 66 | 1.33 | CGC(R) | 10 | 0.53 |
| CUA(L) | 190 | 1.92 | CCA(P) | 107 | 1.95 | CAA(Q) | 84 | 1.75 | CGA(R) | 46 | 2.42 |
| CUG(L) | 55 | 0.56 | CCG(P) | 14 | 0.25 | CAG(Q) | 12 | 0.25 | CGG(R) | 9 | 0.47 |
| AUU(I) | 202 | 1.43 | ACU(T) | 48 | 0.6 | AAU(N) | 53 | 0.73 | AGU(S) | 13 | 0.33 |
| AUC(I) | 81 | 0.57 | ACC(T) | 100 | 1.25 | AAC(N) | 92 | 1.27 | AGC(S) | 44 | 1.11 |
| AUA(M) | 155 | 1.52 | ACA(T) | 159 | 1.99 | AAA(K) | 79 | 1.72 | AGA(*) | 2 | 1.60 |
| AUG(M) | 49 | 0.48 | ACG(T) | 12 | 0.15 | AAG(K) | 13 | 0.28 | AGG(*) | 2 | 1.60 |
| GUU(V) | 59 | 1.07 | GCU(A) | 53 | 0.68 | GAU(D) | 25 | 0.61 | GGU(G) | 28 | 0.46 |
| GUC(V) | 48 | 0.87 | GCC(A) | 129 | 1.66 | GAC(D) | 57 | 1.39 | GGC(G) | 64 | 1.06 |
| GUA(V) | 89 | 1.61 | GCA(A) | 120 | 1.55 | GAA(E) | 79 | 1.65 | GGA(G) | 118 | 1.96 |
| GUG(V) | 25 | 0.45 | GCG(A) | 8 | 0.10 | GAG(E) | 17 | 0.35 | GGG(G) | 31 | 0.51 |
| [1] |
陈慧林, 朱永航, 范海页, 等, 2023. 基于18S和28S rDNA基因的房舍甲螨DNA条形码比较研究[J]. 中国寄生虫学与寄生虫病杂志, 41(2): 163-169.
doi: 10.12140/j.issn.1000-7423.2023.02.006 |
|
doi: 10.12140/j.issn.1000-7423.2023.02.006 |
|
| [2] |
陈治, 高天翔, 2023. 线粒体12S与COI条形码对海洋鱼类的鉴定差异[J]. 海南热带海洋学院学报, 30(2): 10-16.
|
|
|
|
| [3] |
邓家刚, 2008. 广西海洋药物[M]. 南宁: 广西科学技术出版社.
|
|
|
|
| [4] |
董江星, 时伟, 孔晓瑜, 等, 2018. 褐斜鲽(Plagiopsetta glossa)线粒体基因组特征及重排机制研究[J]. 热带海洋学报, 37(1): 1-11.
doi: 10.11978/2017041 |
|
|
|
| [5] |
龚理, 时伟, 司李真, 等, 2013. 鱼类线粒体DNA重排研究进展[J]. 动物学研究, 34(6): 666-673.
|
|
|
|
| [6] |
郭新红, 刘少军, 刘巧, 等, 2004. 鱼类线粒体DNA研究新进展[J]. 遗传学报, 31(9): 983-1000.
|
|
|
|
| [7] |
连总强, 滚双宝, 李力, 等, 2017. 基于第二代测序技术兰州鲇线粒体基因组全序列测定与分析[J]. 水生生物学报, 41(2): 334-345.
|
|
|
|
| [8] |
梁日深, 杨杰銮, 谢瑞琳, 等, 2022. 巨石斑鱼与斜带石斑鱼线粒体基因组测序及物种有效性分析[J]. 中国海洋大学学报, 52(6): 50-61.
|
|
|
|
| [9] |
林龙山, 张静, 宋普庆, 等, 2013. 东山湾及其邻近海域常见游泳动物[M]. 北京: 海洋出版社.
|
|
|
|
| [10] |
刘东, 2005. 中国蛇鳗科鱼类嗅觉器官的比较形态学及其系统发育研究[D]. 上海: 上海海洋大学.
|
|
|
|
| [11] |
刘焕章, 2004. 用mtDNA 12S rRNA序列变异检验鲤形目鱼类系统发育关系[J]. 遗传学报, 31(2): 137-142.
|
|
|
|
| [12] |
刘继兵, 赵洪喜, 2022. 基于18S rRNA的胎毛滴虫感染PCR诊断方法的建立[J]. 中国寄生虫学与寄生虫病杂志, 40(5): 682-685.
doi: 10.12140/j.issn.1000-7423.2022.05.019 |
|
|
|
| [13] |
刘凯, 冯晓宇, 马恒甲, 等, 2020. 钱塘江三角鲂线粒体基因组测序及其结构特征分析[J]. 浙江农业学报, 32(9): 1591-1608.
doi: 10.3969/j.issn.1004-1524.2020.09.08 |
|
doi: 10.3969/j.issn.1004-1524.2020.09.08 |
|
| [14] |
刘文强, 贾玉萍, 赵宏坤, 2006. 16 S rRNA在细菌分类鉴定研究中的应用[J]. 动物医学进展, 27(11): 15-18.
|
|
|
|
| [15] |
刘毅敏, 赵先英, 王祥智, 等, 2006. 基于自由能的生物大分子结构研究[J]. 化学世界, 47(8): 508-510.
|
|
|
|
| [16] |
宁子君, 刘玉萍, 张书飞, 等, 2022. 艾氏蛇鳗线粒体基因组全序列结构分析和系统发育关系探讨[J]. 中国水产科学, 29(9): 1264-1276.
|
|
|
|
| [17] |
钱立富, 2018. 蛇类线粒体基因组结构演化研究及原矛头蝮属生物地理学分析[D]. 合肥: 安徽大学.
|
|
|
|
| [18] |
申欣, 田美, 孟学平, 等, 2014. 鳗鲡目鱼类线粒体蛋白质编码基因易位及系统演化关系分析[J]. 海洋学报, 36(4): 73-81.
|
|
|
|
| [19] |
唐文乔, 张春光, 2004. 蛇鳗科分类综述及中国蛇鳗科系统分类(鱼纲, 鳗鲡目)[J]. 上海水产大学学报, 13(1): 16-22.
|
|
|
|
| [20] |
唐优良, 章群, 余帆洋, 等, 2011. 基于12S rRNA部分序列分析的中国8种笛鲷科鱼类系统发育初探[J]. 海洋科学, 35(2): 22-26.
|
|
|
|
| [21] |
杨婧, 黄原, 2016. 线粒体基因组的高通量测序策略[J]. 生命科学, 28(1): 112-117.
|
|
|
|
| [22] |
张燕萍, 郑红梅, 邵芳, 等, 2016. 沙塘鳢线粒体基因组全序列的测定和分析[J]. 常熟理工学院学报(自然科学), 30(4): 97-104.
|
|
|
|
| [23] |
张稚兰, 林汝榕, 邢炳鹏, 2017. COI基因序列在蛇鳗科鱼类种类鉴定中的适用性研究[J]. 应用海洋学学报, 36(3): 411-416.
|
|
|
|
| [24] |
钟东, 赵贵军, 张振书, 等, 2002. 基因组内碱基分布整体均衡与局部不均衡的研究进展[J]. 遗传, 24(3): 351-355.
|
|
|
|
| [25] |
周慧琦, 2014. 基因组GC含量与碱基、密码子和氨基酸使用偏好的关系[D]. 成都: 电子科技大学.
|
|
|
|
| [26] |
邹新慧, 葛颂, 2008. 基因树冲突与系统发育基因组学研究[J]. 植物分类学报, 46(6): 795-807.
|
|
|
|
| [27] |
|
| [28] |
pmid: 6284948 |
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
doi: 10.1093/nar/gkh340 pmid: 15034147 |
| [34] |
doi: 10.1111/j.1558-5646.1985.tb00420.x pmid: 28561359 |
| [35] |
|
| [36] |
|
| [37] |
doi: 10.1080/10635150390235520 pmid: 14530136 |
| [38] |
doi: 10.1098/rspb.2002.2218 pmid: 12614582 |
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
doi: 10.1093/bioinformatics/17.8.754 pmid: 11524383 |
| [43] |
|
| [44] |
|
| [45] |
|
| [46] |
pmid: 12949142 |
| [47] |
doi: 10.1093/molbev/mst141 pmid: 23955518 |
| [48] |
pmid: 7508516 |
| [49] |
|
| [50] |
pmid: 7608989 |
| [51] |
|
| [52] |
|
| [53] |
doi: 10.1093/nar/25.5.955 pmid: 9023104 |
| [54] |
|
| [55] |
doi: 10.1186/1471-2164-12-84 pmid: 21276253 |
| [56] |
|
| [57] |
|
| [58] |
|
| [59] |
pmid: 12470944 |
| [60] |
|
| [61] |
|
| [62] |
|
| [63] |
|
| [64] |
|
| [65] |
doi: 10.1007/BF00186547 pmid: 7563121 |
| [66] |
doi: 10.1093/molbev/msn083 pmid: 18397919 |
| [67] |
|
| [68] |
|
| [69] |
pmid: 8015429 |
| [70] |
|
| [71] |
doi: 10.1186/1471-2148-13-173 pmid: 23962312 |
| [72] |
|
| [73] |
doi: 10.1093/molbev/msab120 pmid: 33892491 |
| [74] |
|
| [75] |
|
| [76] |
doi: 10.1093/oxfordjournals.molbev.a025661 pmid: 8752002 |
| [77] |
|
| [1] | 霍嘉欣, 李颖心, 宋严, 朱晴, 周伟华, 袁翔城, 黄晖, 刘胜. 木珊瑚科Cladopsammia gracilis和Rhizopsammia wettsteini 线粒体全基因组比较与系统进化分析*[J]. 热带海洋学报, 2024, 43(3): 22-30. |
| [2] | 李能辉, 黄庆, 李航, 曾俊, 吴科锋, 谭华强. 基于形态和分子数据对湛江海域4个江蓠物种(红藻门)的分类学研究[J]. 热带海洋学报, 2024, 43(2): 34-47. |
| [3] | 黄培贤, 姚雪梅, 余巧驰, 张佳玉. 海南澳洲管体星虫线粒体基因组特征及进化分析[J]. 热带海洋学报, 2023, 42(2): 54-63. |
| [4] | 刘巍, 郭海朋, 董鹏生, 燕孟琛, 张德民. 别样玫瑰变色杆菌(Aliiroseovarius sp.)Z3基因组测序及比较基因组分析[J]. 热带海洋学报, 2022, 41(1): 52-61. |
| [5] | 龙超, 罗肇河, 韦章良, 杨芳芳, 李茹, 龙丽娟. 海南三亚鹿回头虫黄藻(Effrenium voratum)的形态学和系统发育学研究*[J]. 热带海洋学报, 2021, 40(4): 35-43. |
| [6] | 徐轶肖, 何喜林, 张腾, 蓝文陆. 北部湾棕囊藻藻华原因种分析[J]. 热带海洋学报, 2020, 39(6): 122-130. |
| [7] | 黄丽芬, 李群, 吕颂辉, 张亮, 谢学东. 中国西沙群岛底栖甲藻热带库里亚藻(Coolia tropicalis)的形态学、系统发育及毒性研究[J]. 热带海洋学报, 2020, 39(3): 86-97. |
| [8] | 江丽春, 李群, 吕颂辉. 广西涠洲岛底栖甲藻前沟藻属(Amphidinium)种类的形态学和系统发育学研究[J]. 热带海洋学报, 2020, 39(3): 106-115. |
| [9] | 董江星, 时伟, 孔晓瑜, 陈世喜. 褐斜鲽(Plagiopsetta glossa)线粒体基因组特征及重排机制研究[J]. 热带海洋学报, 2018, 37(1): 1-11. |
| [10] | 陈斌, 何薇, 李海涛, 吴鹏, 肖瑜璋, 吕向立, 何静. 基于线粒体CO#cod#x02160;基因对南海毛颚动物箭虫科的鉴定及系统发育初探[J]. 热带海洋学报, 2017, 36(6): 100-108. |
| [11] | 司李真, 时伟, 杨敏, 龚理, 孔晓瑜. 硬骨鱼类核糖体基因间隔区的序列特征分析[J]. 热带海洋学报, 2016, 35(6): 74-81. |
| [12] | 范程辉, 刘丽, 沈城, 郭昱嵩. 稀杯盔形珊瑚铜锌超氧化物歧化酶基因全长cDNA 序列的克隆与分析*[J]. 热带海洋学报, 2015, 34(1): 83-89. |
| [13] | 韦蔓新, 范航清, 何本茂, 曹庆先, 李智. 广西铁山港红树林区水体的营养水平与结构特征*[J]. 热带海洋学报, 2013, 32(4): 84-91. |
| [14] | 刘丽, 赵洁, 郭昱嵩, 刘楚吾. 4种笛鲷AFLP引物筛选及其遗传多样性分析[J]. 热带海洋学报, 2012, 31(4): 112-116. |
| [15] | 胡永乐,梁旭方,王琳,李观贵,刘秀霞,王云新,张海发. 斜带石斑鱼胰蛋白酶原和淀粉酶全长cDNA的克隆与序列分析[J]. 热带海洋学报, 2010, 29(5): 125-131. |
|
||